Normalitātes testi
Daudzām statistiskajām analīzēm (parametriskie testi) viens no pieņēmumiem ir, ka dati atbilst normālajam sadalījumam. Programmā R normalitāti datiem var pārbaudīt gan grafiskā veidā (kas šobrīd ir pieņemts veids vismaz daudzās statistikas grāmatās), gan arī analītiski.
Grafiskai normalitātes novērtēšanai izmanto QQ grafikus, ko iegūst ar funkcijas qqnorm() palīdzību, kā argumentu norādot mainīgo (kolonnu), kurai jāpārbauda atbilstība normālajam sadalījumam. Papildus var izmantot arī funkciju qqline(), kas novilks līniju, pēc kuras ir vieglāk novērtēt attēlu. Interpretējot šo grafiku, ir jāskatāt vai grafikā attēlotie punkti apmēram atrodas uz taisnes (vai tuvu tai), kā arī vai nav kādas izteiktas nobīdes no šīs taisnes. Ja punkti atrodas apmēram uz taisnes, tad dati atbilst normālajam sadalījumam.
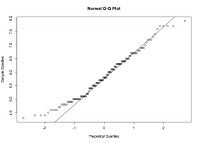
> data(iris)
> attach(iris)
> names(iris)
[1] "Sepal.Length" "Sepal.Width" "Petal.Length" "Petal.Width" "Species"
> qqnorm(Sepal.Length)
> qqline(Sepal.Length)
Analītiskai normalitātes novērtēšanai var izmantot Šapiro testu ar funkciju shapiro.test(), iekavās norādot pārbaudāmo mainīgo (kolonnu). Šajā gadījumā dati atbilst normālajam sadalījumam, ja p vērtība ir lielāka, nekā izvēlētais būtiskuma līmenis.
> shapiro.test(Sepal.Length)
Shapiro-Wilk normality test
data: Sepal.Length
W = 0.9761, p-value = 0.01018
Papildus testiem, kas atrodami programmas pamatversijā, ir specializēta R pakete nortest, kurā ir pieci papildus normalitātes testi. Tā kā katrs no tiem izmanto nedaudz atšķirīgu aprēķināšanas algoritmu, tad iegūtās p vērtības starp testiem atšķiras. Iespējamie testi ir: Anderson-Darling tests (ad.test()), Cramer-von Mises tests (cvm.test()), Lilliefors (Kolmogorov-Smirnov) tests (lillie.test()), Pīrsona hī kvadrāta tests (pearson.test()) un Shapiro-Francia tests (sf.test()).
> library(nortest)
> ad.test(Sepal.Length)
Anderson-Darling normality test
data: Sepal.Length
A = 0.8892, p-value = 0.02251
> cvm.test(Sepal.Length)
Cramer-von Mises normality test
data: Sepal.Length
W = 0.1274, p-value = 0.04706
> lillie.test(Sepal.Length)
Lilliefors (Kolmogorov-Smirnov) normality test
data: Sepal.Length
D = 0.0887, p-value = 0.005788
> pearson.test(Sepal.Length)
Pearson chi-square normality test
data: Sepal.Length
P = 17.4, p-value = 0.1352
> sf.test(Sepal.Length)
Shapiro-Francia normality test
data: Sepal.Length
W = 0.9796, p-value = 0.02621
